Traditional Nearest Neighbors Algorithms
Introduce to kNN(k Neareset Neighbors)
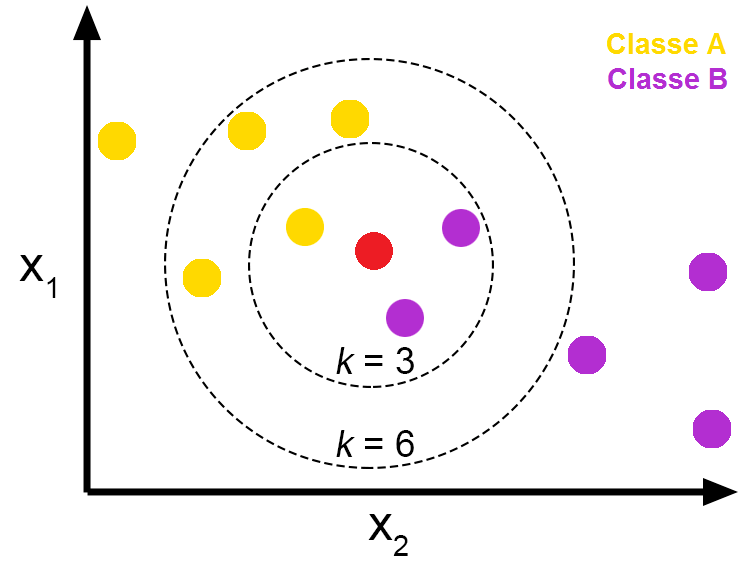
- 거리가 가장 가까운 이웃 k개를 반환하는 알고리즘
- 혹은 가장 인접한 벡터 k개를 선택하여 voting을 통해 classification
- Distance Measurement로는 크게 3가지 존재
- L1 norm(Manhattan Distance)
- 2차원: $D=\left\vert x_1-x_2 \right\vert+\left\vert y_1-y_2 \right\vert$
- 3차원: $D=\left\vert x_1-x_2 \right\vert+\left\vert y_1-y_2 \right\vert+\left\vert z_1-z_2\right\vert$
- L2 norm(Euclidean Distance)
- 2차원: $D=\sqrt{(x_1-x_2)^2+(y_1-y_2)^2}$
- 3차원: $D=\sqrt{(x_1-x_2)^2+(y_1-y_2)^2+(z_1-z_2)^2}$
- Cosine Distance
- $1-\frac{X \cdot Y}{\left\vert X \right\vert _2 \cdot \left\vert Y \right\vert _2}$
- L1 norm(Manhattan Distance)
k Nearest Neighbors Algorithms
Brute-Force
- 특징
- 모든 데이터포인트 쌍의 거리를 계산 (pair-wise distance)
- 저차원, 소규모 데이터셋에 대해 효과적
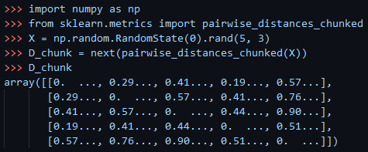
- Time Complexity
- Dimension: $D$
- Number of Vectors: $N$
- pair-wise distance 연산: $O(DN^2)$
- pair-wise distance sorting: $O(N^2logN)$
- Sorted List에서 top k개 반환: $O(1)$
- 최종 Time Complexity: $O(DN^2+N^2logN)$
요약
- Time Complexity만 보아도 어마무시하다. 느리다.
K-D Tree
- 특징
- kNN 개념과는 달리 K-D 트리를 미리 구축
- 구축된 트리에서 query point가 어느 리프 노드에 위치하는지 탐색
- 후에 트리를 순회하며 거리를 비교
- K-D Tree
- k-D Tree: k차원 공간(k-Dimension)의 점들을 구조화 하는 공간 분할 자료구조
- 중심 데이터포인트를 기준으로 분할 초평면을 만들어 트리 생성
- BST(Binary Search Tree)와 유사하나, k-Dimension 상의 데이터를 트리로 구축할 수 있음
- BST를 x축에 대해서 수행 -> y축에 대해서 수행 -> z축에 대해서 수행 -> x축에 대해서 수행 -> y축에 대해서 …
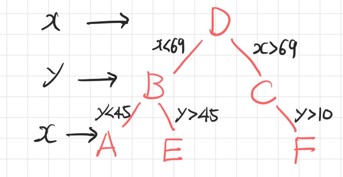
k-D Tree Construction Algorithm
- Precondition
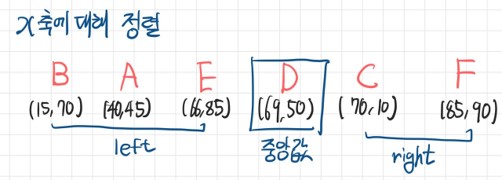
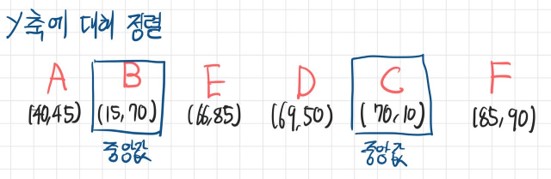
- 2차원 실수 공간 (X측, Y축)
- X축에 대해 정렬한 후 중앙값을 기준으로 배열 분할
- Y축에 대해 먼저 정렬하여 분할할 수도 있음
- 이는 각 축의 분산에 의해 결정됨
- Y축에 대해 정렬한 후 중앙값을 기준으로 배열 분할
- 위 과정을 반복하면서 트리 생성
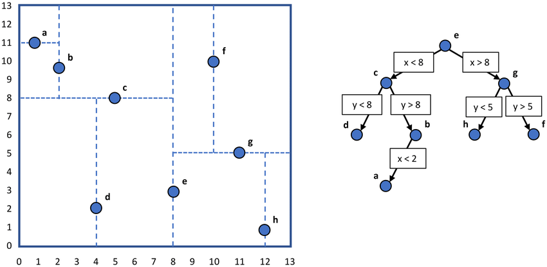
K-D Tree Nearest Neighbor Search Algorithm
- 새로운 점이 트리의 어디에 삽입되어야 하는지 탐색
- 도달한 리프 노드를 기준으로 자신이 속한 subtree를 우선적으로 탐색
- 자신이 속한 subtree에서 k개의 최근접 이웃을 모두 탐색하지 못한 경우, 부모 노드의 subtree에서 순회
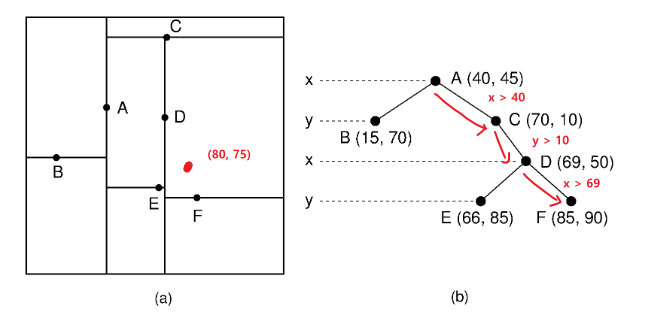
Time Complexity
- Time Complexity(Notations)
- Dimension: $D$
- Number of Vectors: $N$
- Time Complexity(Building)
- 추가할 point가 어느 subtree에 위치하는지 탐색: $O(DlogN)$
- 총 벡터 n개를 만족할 때가지 트리 구축: $O(N)$
- 최종 Time Complexity: $O(DNlogN)$
- Time Complexity(Search)
- 평균적인 복잡도
- 추가할 point가 어느 subtree에 위치하는지 탐색: $O(DlogN)$
- 불균형한 트리인 경우, i.e. Skewed Tree
- 추가할 point가 어느 subtree에 위치하는지 탐색: $O(DN)$
- 평균적인 복잡도
요약
- 특정 차원의 데이터가 불균형한 경우, 해당 차원에서 skewed될 가능성이 있음
- 이는 k-D Tree의 성능을 크게 저하시킴
- 고차원 데이터에서 성능 하락의 주요 원인
Ball Tree
- 특징
- k-D 트리와 마찬가지로 트리를 미리 구축하여 트리 순회를 통해 nearest neighbor 탐색
- 경계를 정할 때, k-D 트리는 hyper plane이었다면, Ball Tree는 hyper sphere
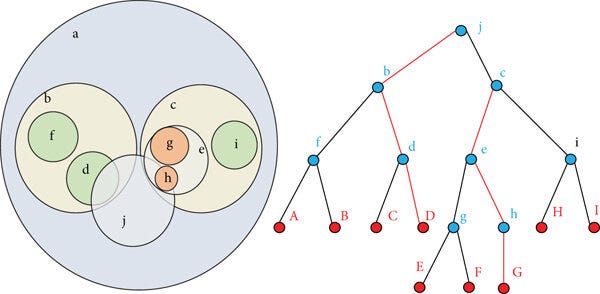
Ball Tree Construction
- 중심점 계산: 주어진 데이터 포인트 집합에 대해 중심점을 계산(각 차원 별 평균 혹은 무게중심(centroid))
- 반경 결정: 중심점으로부터 가장 먼 점까지의 거리를 반경으로 설정
- 분할: 중심점을 기준으로 데이터 포인트들을 두 개의 하위 집합으로 분할. 이를 재귀적으로 반복
- 두 개의 시드 포인트 선택
- 주어진 데이터 집합 $P$에서 가장 먼 두 점을 시드(seed)로 선택. 이 점을 $s_1$, $s_2$라 하자.
- 시드 포인트 $s_1$과 $s_2$는 각 하위 구의 초기 중심점 역할
- 할당
- 나머지 데이터 포인트들을 두 시드 포인트 $s_1$과 $s_2$ 중 가까운 쪽에 할당
- 하위 구의 중심점과 반경 계산
- 각 하위 집합 $P_1$과 $P_2$에 대해 새로운 중심점과 반경 계산
- 중심점 $C_1$과 $C_2$
- $C_1$은 $P_1$의 중심점, $C_2$는 $P_2$의 중심점
- 반경 $R_1$과 $R_2$
- $R_1$은 $P_1$내에서 중심점 $C_1$으로부터 가장 먼 데이터포인트까지의 거리
- $R_2$은 $P_2$내에서 중심점 $C_2$으로부터 가장 먼 데이터포인트까지의 거리
- 두 개의 시드 포인트 선택
Time Complexity
- Time Complexity(Notations)
- Dimension: $D$
- Number of Vectors: $N$
- Time Complexity(Building)
- 추가할 point가 어느 subtree에 위치하는지 탐색: $O(DlogN)$
- 총 벡터 n개를 만족할 때가지 트리 구축: $O(N)$
- 최종 Time Complexity: $O(DNlogN)$
- Time Complexity(Search)
- 평균적인 복잡도
- 추가할 point가 어느 subtree에 위치하는지 탐색: $O(DlogN)$
- 불균형한 트리인 경우, i.e. Skewed Tree
- 추가할 point가 어느 subtree에 위치하는지 탐색: $O(DN)$
- 평균적인 복잡도
요약
- k-D 트리에 비해 균형 잡힌 트리를 생성할 가능성이 높음
- 차원 별 밀도 편향에 따른 skewed는 해결하였으나, 전체적인 밀도 편향에 따른 skewed 존재
- 고차원 공간에서 효과적인 분할이 어려워짐
Appendix (scikit-learn source code)
brute-force kNN
# Joblib-based backend, which is used when user-defined callable
# are passed for metric.
# This won't be used in the future once PairwiseDistancesReductions
# support:
# - DistanceMetrics which work on supposedly binary data
# - CSR-dense and dense-CSR case if 'euclidean' in metric.
# for efficiency, use squared euclidean distances
if self.effective_metric_ == "euclidean":
radius *= radius
kwds = {"squared": True}
else:
kwds = self.effective_metric_params_
reduce_func = partial(
self._radius_neighbors_reduce_func,
radius=radius,
return_distance=return_distance,
)
chunked_results = pairwise_distances_chunked(
X,
self._fit_X,
reduce_func=reduce_func,
metric=self.effective_metric_,
n_jobs=self.n_jobs,
**kwds,
)
if return_distance:
neigh_dist_chunks, neigh_ind_chunks = zip(*chunked_results)
neigh_dist_list = sum(neigh_dist_chunks, [])
neigh_ind_list = sum(neigh_ind_chunks, [])
neigh_dist = _to_object_array(neigh_dist_list)
neigh_ind = _to_object_array(neigh_ind_list)
results = neigh_dist, neigh_ind
else:
neigh_ind_list = sum(chunked_results, [])
results = _to_object_array(neigh_ind_list)
if sort_results:
for ii in range(len(neigh_dist)):
order = np.argsort(neigh_dist[ii], kind="mergesort")
neigh_ind[ii] = neigh_ind[ii][order]
neigh_dist[ii] = neigh_dist[ii][order]
results = neigh_dist, neigh_ind
k-D Tree, Ball Tree
cdef int allocate_data(
BinaryTree tree,
intp_t n_nodes,
intp_t n_features,
) except -1:
"""Allocate arrays needed for the KD Tree"""
tree.node_bounds = np.zeros((2, n_nodes, n_features), dtype=)
return 0
cdef int init_node(
BinaryTree tree,
NodeData_t[::1] node_data,
intp_t i_node,
intp_t idx_start,
intp_t idx_end,
) except -1:
"""Initialize the node for the dataset stored in tree.data"""
cdef intp_t n_features = tree.data.shape[1]
cdef intp_t i, j
cdef float64_t rad = 0
cdef * lower_bounds = &tree.node_bounds[0, i_node, 0]
cdef * upper_bounds = &tree.node_bounds[1, i_node, 0]
cdef const * data = &tree.data[0, 0]
cdef const intp_t* idx_array = &tree.idx_array[0]
cdef const * data_row
# determine Node bounds
for j in range(n_features):
lower_bounds[j] = INF
upper_bounds[j] = -INF
# Compute the actual data range. At build time, this is slightly
# slower than using the previously-computed bounds of the parent node,
# but leads to more compact trees and thus faster queries.
for i in range(idx_start, idx_end):
data_row = data + idx_array[i] * n_features
for j in range(n_features):
lower_bounds[j] = fmin(lower_bounds[j], data_row[j])
upper_bounds[j] = fmax(upper_bounds[j], data_row[j])
for j in range(n_features):
if tree.dist_metric.p == INF:
rad = fmax(rad, 0.5 * (upper_bounds[j] - lower_bounds[j]))
else:
rad += pow(0.5 * abs(upper_bounds[j] - lower_bounds[j]),
tree.dist_metric.p)
node_data[i_node].idx_start = idx_start
node_data[i_node].idx_end = idx_end
# The radius will hold the size of the circumscribed hypersphere measured
# with the specified metric: in querying, this is used as a measure of the
# size of each node when deciding which nodes to split.
node_data[i_node].radius = pow(rad, 1. / tree.dist_metric.p)
return 0
참고 문헌
- cjkangme.log. “[3D] KD Tree와 BVH” 2024.01.11. https://velog.io/@cjkangme/3D-KD-Tree%EC%99%80-BVH
- 고귀양이의 노트. “ball tree” 2019.08.01. https://nobilitycat.tistory.com/entry/ball-tree
- Geetha Mattaparthi. “Ball tree and KD Tree Algorithms” 2024.01.23. https://medium.com/@geethasreemattaparthi/ball-tree-and-kd-tree-algorithms-a03cdc9f0af9
- “1.6 Nearest Neighbors”. Scikit Learn. Version 1.4.1. https://scikit-learn.org/stable/modules/neighbors.html
- “scikit-learn/sklearn/neighbors”. https://github.com/scikit-learn/scikit-learn/tree/main/sklearn/neighbors
- “scikit-learn/sklearn/tree/_tree.pyx”. https://github.com/scikit-learn/scikit-learn/blob/main/sklearn/tree/_tree.pyx


댓글남기기